BIB | 临港实验室合作开发空间转录组空间域识别算法MCGAE:深度学习整合多模态信息, 助力疾病机制和药靶发现研究
在多细胞组织的研究中,为了深入理解生物过程和分子动态,必须在空间背景下进行高分辨率的多模态数据整合。空间转录组学技术的进展虽然极大提升了基因表达在空间上的解析能力,但传统分析方法往往忽略了空间关系和图像信息的融合,这在复杂组织环境的解析中存在局限性。现有的图神经网络方法虽然引入了空间位置的邻接矩阵,提升了空间域识别的精度,但多数方法依赖于预定义的相似性度量,难以适应不同数据集间的多样性。此外,现有方法在使用图像信息方面效率较低,无法充分捕捉细胞空间分布中的形态学特征。
2024年11月22日,临港实验室魏武研究团队在 Briefings in Bioinformatics 期刊上在线发表了题为“MCGAE: unraveling tumor invasion through integrated multimodal spatial transcriptomics”的研究论文。研究团队开发了多视图对比图自编码器(Multi-View Contrastive Graph Autoencoder, MCGAE),这是一种专为空间转录组数据分析设计的深度学习框架。MCGAE通过结合基因表达、空间坐标和图像特征,创建多模态、多视图的生物表征,显著提升了空间域识别的准确性。该框架利用对比图卷积网络与注意力机制,分别生成特定模态的空间表示并整合为统一的嵌入,有效增强了噪声环境下的鲁棒性。MCGAE支持多种视图结构的构建,即便在缺少图像信息的情况下,仍能通过基因和空间数据实现精准的空间域划分。该方法在多种真实及模拟数据集上展示出优异表现,尤其在肿瘤数据中展现了对肿瘤区域的精确识别与分子调控特征的深度解析,为复杂组织、疾病机制研究和药物靶点发现提供了强有力的工具。

MCGAE在多种数据集上进行了广泛的测试,包括10x Visium、Stereo-seq、Slide-seq、STARmap、MERFISH数据集,以及模拟数据集。测试结果表明,MCGAE在精确度和鲁棒性方面显著优于十一种现有算法。尤其是在结合肿瘤影像数据时,MCGAE在空间域识别和聚类精度上有了明显提升。此外,通过数据去噪、识别空间可变基因(SVGs)以及提取三维空间域特征,MCGAE实现了对肿瘤侵袭区域的深入探索,从而促进了肿瘤医学研究。
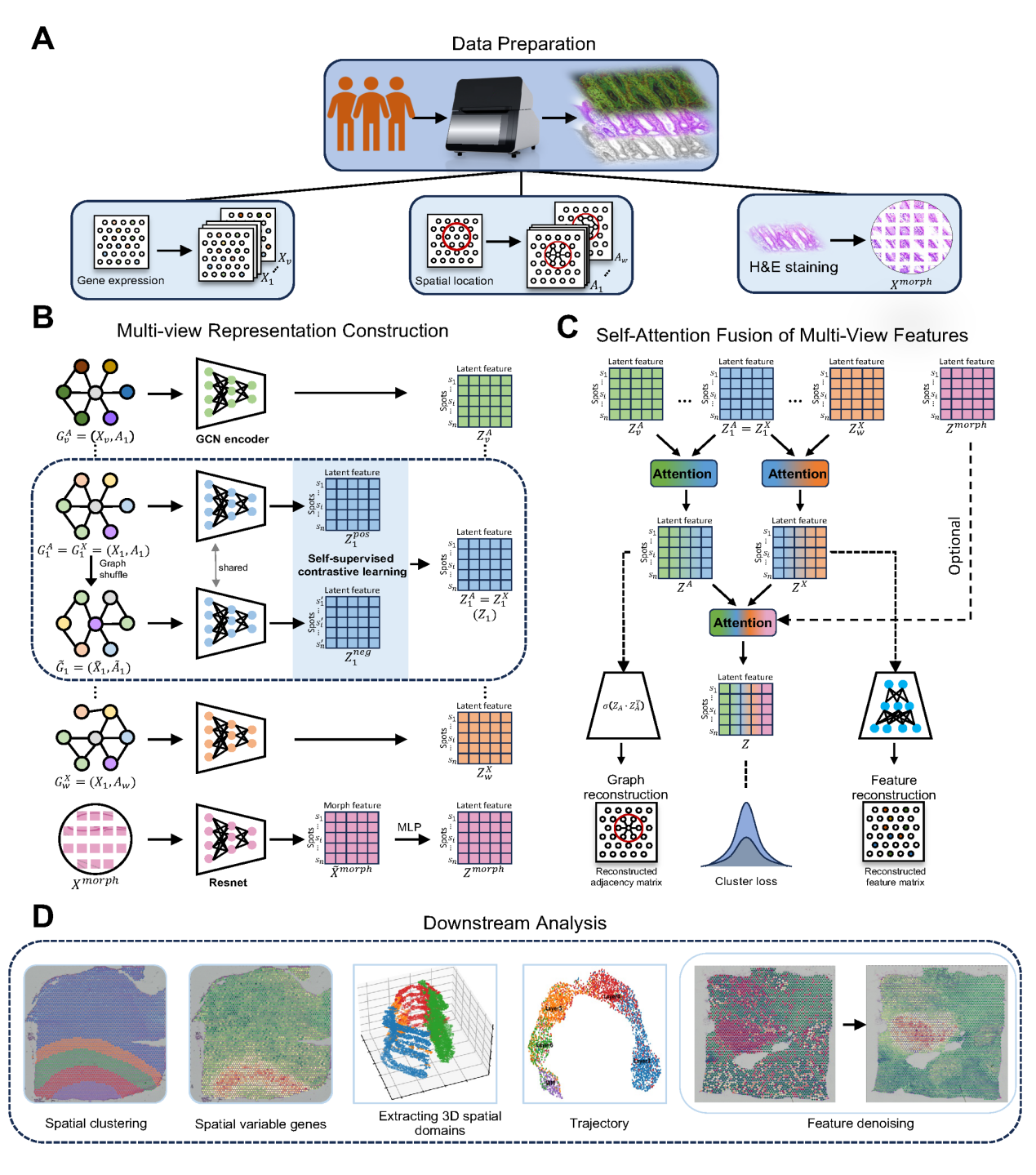
图1. MCGAE研究设计示意图及下游应
在空间多模态转录组数据的背景下,MCGAE首先获取原始基因表达矩阵和根据空间坐标计算的邻接矩阵,并采用模块化建模的方法,使得用户可以灵活选择不同的增强方法,例如简单的自编码器等,以获得基因表达矩阵X的增强视图。此外,通过多种相似性度量的使用,MCGAE能够构建邻接矩阵A的多重视图,丰富数据分析的深度,捕捉空间转录组数据中固有的多重关系和信息维度。
在多视图构建阶段,MCGAE通过对比图卷积神经网络(Graph Convolutional Network, GCN)和注意力模块获取特定模态的空间点位表示。具体而言,MCGAE固定X1并将其与不同的A视图组合,每对(X1,Aj)通过图卷积网络进行处理,从而提取与X1相关的多视图表示。这些表示通过注意力机制融合成一个综合嵌入ZX,用于重构原始的X1。同样地,通过保持A1不变并变换Xi,MCGAE获得了A1的视图特异性表示,并将这些表示聚合为ZA。其中(X1,A1)被指定为基础图,代表了最初的表达数据。通过自监督对比学习,进一步优化这些生物学表示的准确性。在形态图像可用的情况下,MCGAE采用预训练的ResNet50模型提取图像特征,从而获得图像嵌入Zmorph,增强模型在处理多模态数据方面的能力。
在最终融合阶段,MCGAE通过注意力机制将ZX、ZA和Zmorph融合为最终的综合嵌入Z。这个嵌入进一步通过无监督深度迭代聚类策略进行优化,以增强表征的聚合度,并应用于下游的分析任务,如空间域识别、数据去噪、空间变异基因(SVGs)识别、轨迹推断和三维空间域提取等。通过集成多视图对比图神经网络、注意力机制以及深度迭代聚类,MCGAE有效增强了噪声的鲁棒性和空间特异性。相比于传统单视图的图神经网络模型,MCGAE在多视角图构建和自监督学习的协同下,大幅度提升了空间转录组数据的分析精度和适用性。
总之,MCGAE的提出不仅丰富了空间转录组学的多模态数据处理方法,也为生物医学研究提供了新的工具。通过将多视图对比学习、注意力机制和深度聚类相结合,MCGAE显著增强了对空间结构和基因表达模式的解析能力,为未来的空间转录组学和疾病研究提供了强大支持。这一方法有望在组织异质性研究、肿瘤微环境分析以及临床医学中的复杂组织病理分析中发挥更大作用,进一步推动个性化医疗和创新药物的研发。
临港实验室魏武研究员,中科院生化所陈洛南研究员,国科大杭州高等研究院张传超助理研究员为本文通讯作者,临港实验室助理研究员杨逸文、东京大学张城铭博士为本文共同第一作者。本研究得到国家重点研发计划、国家自然科学基金等项目的资助。
附件下载:



