Cell Discovery | 临港实验室合作开发整合内源因素的深度学习模型提高碱基编辑预测准确性
单碱基编辑技术自问世以来,因其高效性和特异性备受广泛关注及应用。为方便单碱基编辑器的应用,已有科研工作者通过文库的方式将sgRNA与靶位点共同随机整合到基因组的方式,高通量研究碱基编辑器在不同位点的编辑效果。但该方法存在极大的局限性,只能研究sgRNA序列本身对编辑效果的影响,而无法研究基因组表观信息对其影响。
2月20日,临港实验室魏武研究团队联合中国农业科学院深圳农业基因组研究所左二伟研究团队、中国科学院脑科学与智能技术卓越创新中心孙怡迪研究团队在 Cell Discovery 在线发表了题为“Deep learning models incorporating endogenous factors beyond DNA sequences improve the prediction accuracy of base editing outcomes”的研究论文。通过系统比较碱基编辑器在外源整合靶位点及基因组内源靶位点的编辑效果差异,研究人员发现碱基编辑效果不仅受靶位点序列影响,而且也受靶位点区域的DNA甲基化、组蛋白修饰等表表观因素的影响,在此基础上开发了可准确预测腺嘌呤单碱基编辑器ABE和胞嘧啶单碱基编辑器CBE在基因组内源靶位点编辑效果的预测模型BE_Endo(http://www.sunlab.fun:3838/BE_Endo)。BE_Endo模型的建立极大方便在研究过程中引导sgRNA的筛选,对进一步指导基因编辑工具的应用具有重要意义。

为进一步查明基因组表观信息,如DNA甲基化、组蛋白修饰、染色体可及性等因素对单碱基编辑器编辑效果的影响。研究人员首先设计针对基因组不同区域的sgRNA, 并同时将其构建到外源文库中,进一步检测单碱基编辑器在基因组内源靶位点及外源整合靶位点的编辑效果的差异。研究结果表明,单碱基编辑器在内源靶位点及外源整合靶位点的编辑效率只有中度的相关性(图1),并且在序列偏好性上呈现极大的差异。进一步分析发现编辑效果明显受到靶位点区域的染色体可及性、DNA甲基化、组蛋白修饰等表观因素的影响。在充分分析编辑效果差异与基因组表观信息相关性的基础上,研究人员开发了可高效、准确预测其在内源靶位点编辑效果的深度学习模型BE_Endo,这为后续其他科研人员利用单碱基编辑器开展研究工作提供极大了方便(图2)。
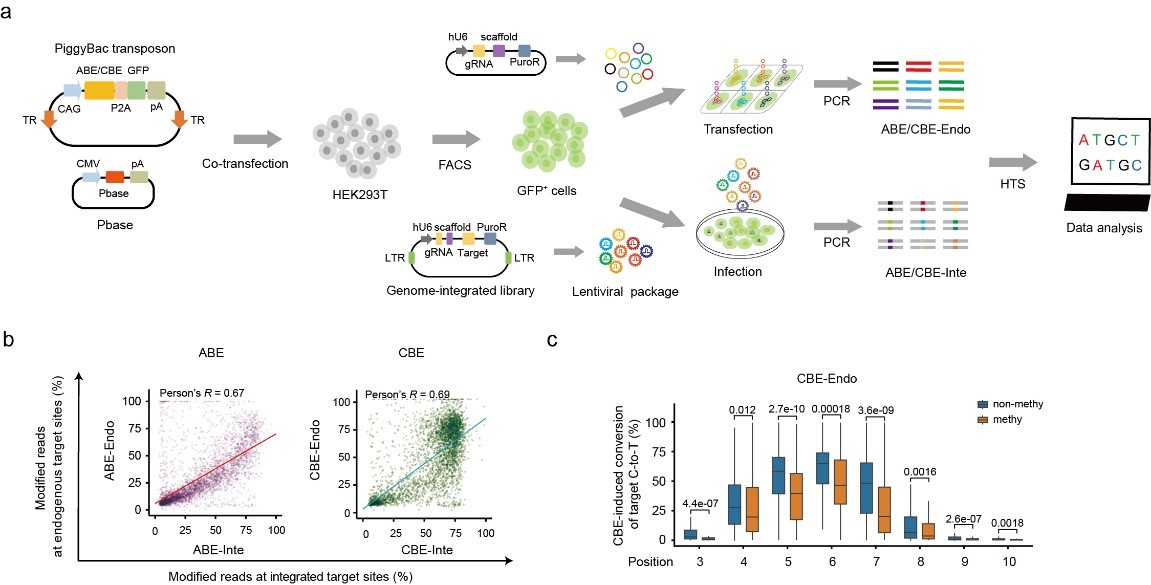
图1. 实验流程及单碱基编辑器在内源位点及整合位点编辑差异
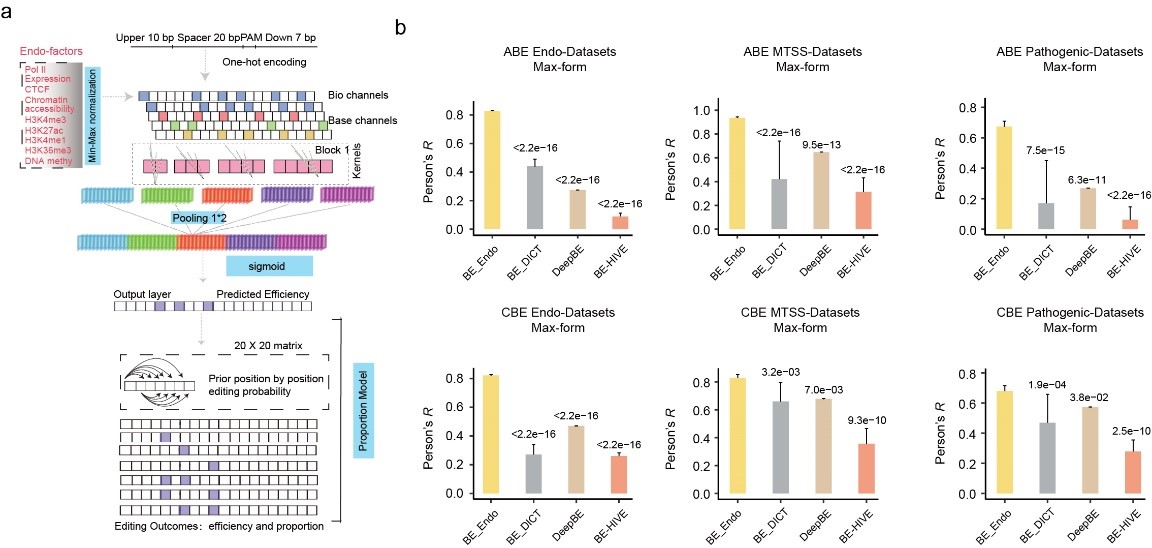
图2. 深度学习模型的建立及不同模型预测效果的比较
中国农业科学院深圳农业基因组研究所副研究员袁堂龙、中国科学院脑科学与智能技术卓越创新中心博后吴垒磊、中国科学院上海营养与健康研究所博士生李世燕、广西大学博士生郑基坛、华中农大博士生李娜娜、华中农大硕士生肖潇为本论文的共同第一作者。中国农业科学院深圳农业基因组研究所左二伟研究员、中国科学院脑科学与智能技术卓越创新中心孙怡迪研究员及临港实验室魏武研究员为本文的共同通讯作者。
该研究得到了国家自然科学基金、国家重点研发计划、中国农业科学院科技创新工程、中国博士后科学基金会、广东省基础与应用基础研究基金等资助。
附件下载:



